简单blast比对:FAD4-like基因在大豆、拟南芥和芝麻中的比对与进化关系研究
01 研究背景
芝麻油脂含量高,应该存在某一基因控制其油脂合成,将大豆(Glycine
max)、拟南芥(Arabidopsis thaliana)、芝麻(Sesamum
indicum)的序列进行比对,寻找芝麻中与油脂合成相关的序列,FAD4-like基因在油脂合成通路中比较重要,因此本次研究芝麻、大豆、拟南芥中FAD4-like基因的进化关系。
02 分析
(1)工作目录
创建文件夹,并把资料存到工作文件夹FAD4-like中
1
2
3
4
5
6
7
8
9
10
| mkdir FAD4-like
less -S
cp /home/ubuntu/work/rnaseq-apple-training/linux-basic/DATA/atha.fasta FAD4-like/atha.fasta
less -S atha.fasta
cp /home/ubuntu/work/rnaseq-apple-training/linux-basic/DATA/atha_FAD4.fa atha_FAD4.fa
less -S atha_FAD4.fa
cp /home/ubuntu/work/rnaseq-apple-training/linux-basic/DATA/sind.fasta ./
cp /home/ubuntu/work/rnaseq-apple-training/linux-basic/DATA/gmax.fasta ./
cat atha.fasta gmax.fasta sind.fasta >all.fasta
|
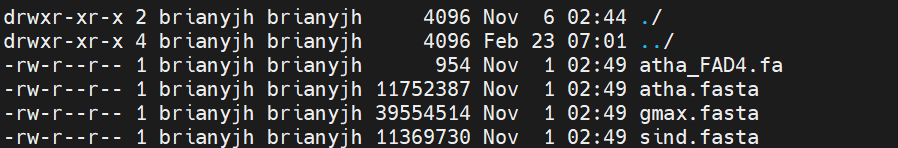
文件夹FAD4-like有以下文件:
(2)参考基因序列
从油脂基因数据库中寻找拟南芥FAD4-like基因(因为拟南芥被研究得比较透彻)
拟南芥FAD4-like基因有三条,At1g62190、At2g22890、At4g27030
将蛋白序列存放在 atha_FAD4.fa 中作为 拟南芥query
蛋白序列
 拟南芥query序列
拟南芥query序列
查看拟南芥中3条FAD4-like基因蛋白序列
1
2
3
4
5
6
7
8
| (base) brianyjh@debian:~/rnaseq_data/rnaseq-apple-training/FAD4-like$ cat atha_FAD4.fa
>atha|AT1G62190.1
MAVSFQTKNPLRPITNIPRSYGPTRVRVTCSVTTTNPQLNHENLVVEKRLVNPPLSKNNDPTLQSTWTHRLWVAAGSTTIFASFAKSIIGGFGSHLWLQPALACYAGYVFADLGSGVYHWAIDNYGGASTPIVGAQLEASQGHHKYPWTITKRQFANNSYTIARAITFIVLPLNLAINNPLFHSFVSTFAFCILLSQQFHAWAHGTKSKLPPLVMALQDMGLLVSRKDHPGHHQAPYNSNYCVVSGAWNKVLDESNLFKALEMALFFQFGVRPNSWNEPNSDWTEETETNFFTKI
>atha|AT2G22890.1
MATSLQTKYTLNPITNNIPRSHRPSFLRVTSTTNSQPNHEMKLVVEQRLVNPPLSNDPTLQSTWTHRLWVAAGCTTVFVSFSKSIIGAFGSHLWLEPSLAGFAGYILADLGSGVYHWATDNYGDESTPLVGIHIEDSQDHHKCPWTITKRQFANNLHFMARGTTLIVLPLDLAFDDHVVHGFVSMFAFCVLFCQLFHAWAHGTKSKLPPLVVGLQDIGLLVSRIHHMNHHRAPYNNNYCVVSGVWNKVLDESNVFKAMEMVLYIQLGVRPRSWTEPNYE
>atha|AT4G27030.1
MAVSLPTKYPLRPITNIPKSHRPSLLRVRVTCSVTTTKPQPNREKLLVEQRTVNLPLSNDQSLQSTKPRPNREKLVVEQRLASPPLSNDPTLKSTWTHRLWVAAGCTTLFVSLAKSVIGGFDSHLCLEPALAGYAGYILADLGSGVYHWAIDNYGDESTPVVGTQIEAFQGHHKWPWTITRRQFANNLHALAQVITFTVLPLDLAFNDPVFHGFVCTFAFCILFSQQFHAWAHGTKSKLPPLVVALQDMGLLVSRRQHAEHHRAPYNNNYCIVSGAWNNVLDESKVFEALEMVFYFQLGVRPRSWSEPNSDWIEETEISNNQA
|
注:这里的序列id的写法:AT4G27030.1中AT4G27030是基因id,“.1”是转录本
此时已有拟南芥FAD4-like基因,如果从大豆或芝麻中找到对应相似的基因,常见两种方法:
一个是基于blast比对,一个是基于结构域,常常二者结合比较严谨
这里使用blast比对将拟南芥的FAD4基因家族分别与大豆和芝麻中做比对,寻找匹配度比较高的序列
(3)blast数据库构建
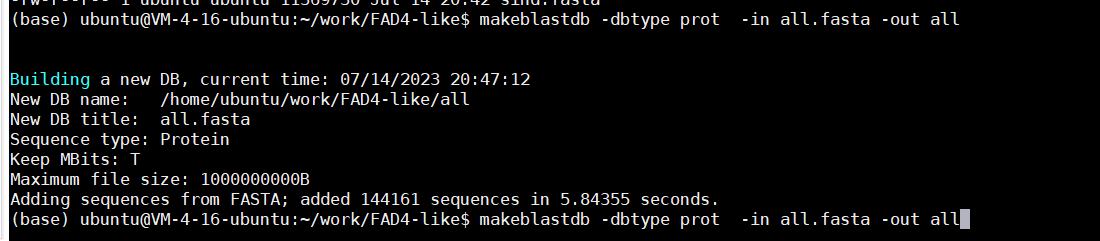
1.首先构建blast数据库
1
| makeblastdb -dbtype prot -in all.fasta -out all
|
-dbtype 数据类型,核酸或者蛋白prot
这里all.fasta也包含拟南芥序列,已经验证后续blast比对的参数是正确的。
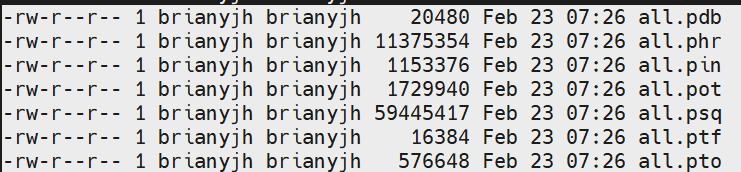
得到结果,生成一些数据库文件
(4)blast比对
1
| blastp -query atha_FAD4.fa -db all -out blast.out -evalue 1e-20 -outfmt 7
|
-query 输入数据
-db 把刚才生成的数据库名称写上(这里是all
-evalue 把e-value设置成\(1^{-10}\)
-outfmt 7 = Tabular with comment lines,
带注释信息的表格
查看比对结果
结果:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
|
atha|AT1G62190.1 atha|AT1G62190.1 100.000 295 0 0 1 295 1 295 0.0 613
atha|AT1G62190.1 atha|AT4G27030.1 68.770 317 70 2 1 288 1 317 1.82e-160 451
atha|AT1G62190.1 atha|AT2G22890.1 70.775 284 76 4 1 282 1 279 2.33e-144 408
atha|AT1G62190.1 gmax|KRG98807 54.861 288 121 1 11 289 15 302 2.15e-113 331
atha|AT1G62190.1 gmax|KRH46136 55.160 281 106 3 27 289 28 306 2.10e-111 326
atha|AT1G62190.1 sind|SIN_1014454 50.336 298 120 4 1 290 1 278 6.80e-102 301
atha|AT1G62190.1 sind|SIN_1005002 47.737 243 122 1 51 288 10 252 1.26e-79 243
atha|AT1G62190.1 sind|SIN_1005003 51.598 219 105 1 65 282 26 244 1.33e-76 235
atha|AT1G62190.1 sind|SIN_1004729 42.060 233 104 2 56 283 16 222 1.56e-57 186
atha|AT1G62190.1 sind|SIN_1005004 47.794 136 70 1 147 282 74 208 2.89e-35 127
atha|AT2G22890.1 atha|AT2G22890.1 100.000 279 0 0 1 279 1 279 0.0 582
atha|AT2G22890.1 atha|AT4G27030.1 68.269 312 65 3 1 279 1 311 9.74e-152 428
atha|AT2G22890.1 atha|AT1G62190.1 70.775 284 76 4 1 279 1 282 2.20e-144 408
atha|AT2G22890.1 gmax|KRG98807 59.925 267 96 2 24 279 29 295 5.88e-113 329
atha|AT2G22890.1 gmax|KRH46136 56.000 300 110 4 1 279 1 299 3.70e-112 327
atha|AT2G22890.1 sind|SIN_1014454 56.627 249 84 3 36 279 41 270 3.24e-97 288
atha|AT2G22890.1 sind|SIN_1005002 49.784 231 111 2 54 279 16 246 1.17e-75 232
atha|AT2G22890.1 sind|SIN_1005003 48.148 216 111 1 62 276 26 241 2.15e-71 221
atha|AT2G22890.1 sind|SIN_1004729 40.179 224 103 3 61 279 24 221 8.03e-52 171
atha|AT2G22890.1 sind|SIN_1005004 44.853 136 74 1 144 279 74 208 5.89e-32 119
atha|AT4G27030.1 atha|AT4G27030.1 100.000 323 0 0 1 323 1 323 0.0 672
atha|AT4G27030.1 atha|AT1G62190.1 68.652 319 67 3 1 317 1 288 2.32e-159 448
atha|AT4G27030.1 atha|AT2G22890.1 68.269 312 65 3 1 311 1 279 1.13e-151 428
atha|AT4G27030.1 gmax|KRG98807 62.126 301 94 3 19 317 19 301 7.04e-133 382
atha|AT4G27030.1 gmax|KRH46136 64.906 265 80 1 66 317 41 305 3.69e-130 375
atha|AT4G27030.1 sind|SIN_1014454 57.994 319 85 9 1 315 1 274 9.96e-118 342
atha|AT4G27030.1 sind|SIN_1005002 52.586 232 105 2 86 312 16 247 4.65e-84 255
atha|AT4G27030.1 sind|SIN_1005003 54.751 221 95 2 94 311 26 244 2.17e-78 241
atha|AT4G27030.1 sind|SIN_1004729 42.222 225 99 2 93 312 24 222 6.35e-57 185
atha|AT4G27030.1 sind|SIN_1005004 53.788 132 60 1 180 311 78 208 5.63e-38 135
|
(5)提取结果
1
2
3
4
5
| grep -v "#" blast.out >blast2.out
awk '$3>50 && $11<1e-30 {print $2}' blast2.out >blast3.out
grep -v "#" blast.out | awk '$3>50 && $11<1e-30{print $2}' |sort|uniq >protein_ids.txt
|
1
| seqtk subseq all.fasta protein_ids.txt > FAD4-like.fasta
|
seqtk subseq [options]<in.fa> <in.bed>
<name.list>
到FileZilla把文件下载下来
然后放到MEGA里面进行比对
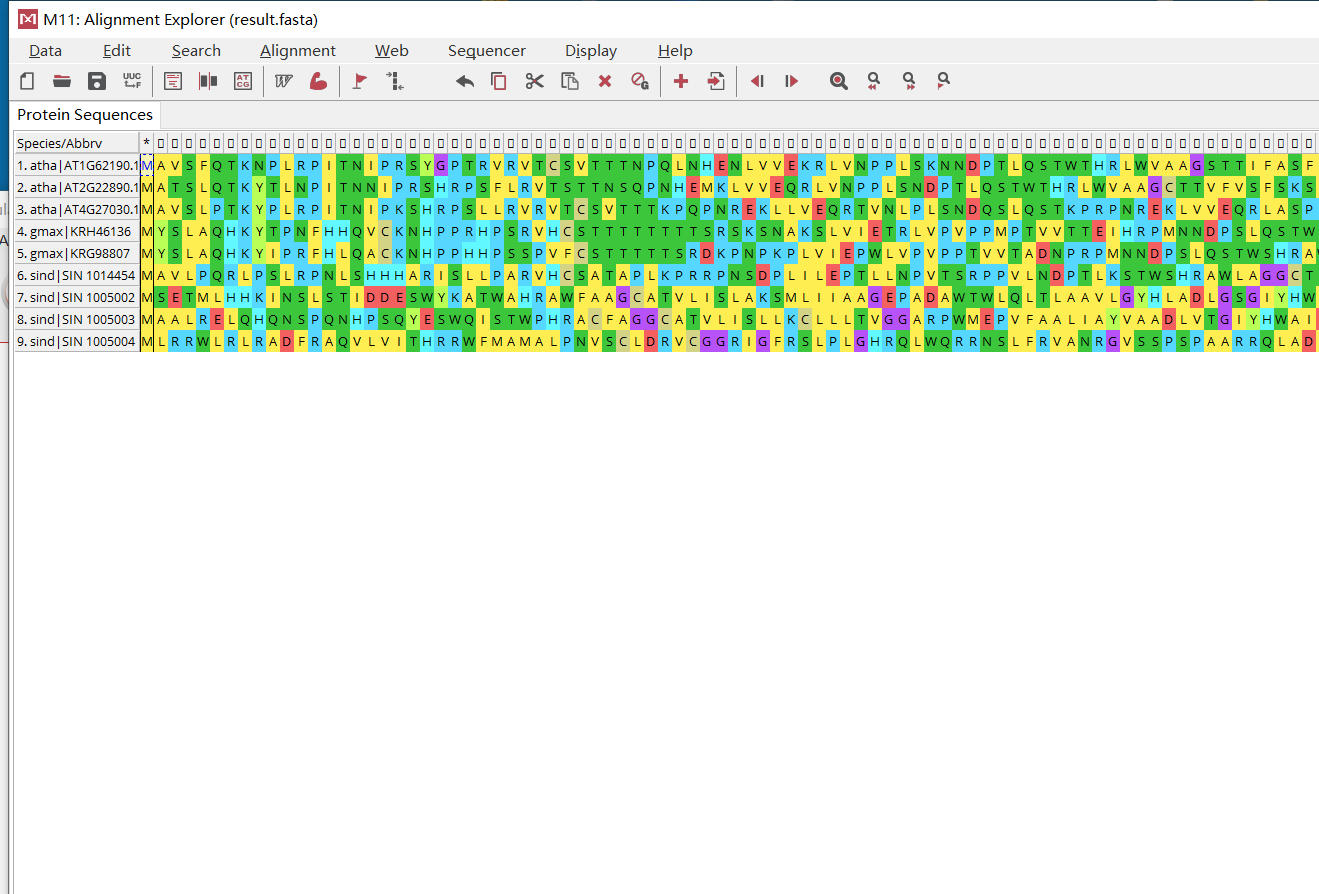
多序列比对
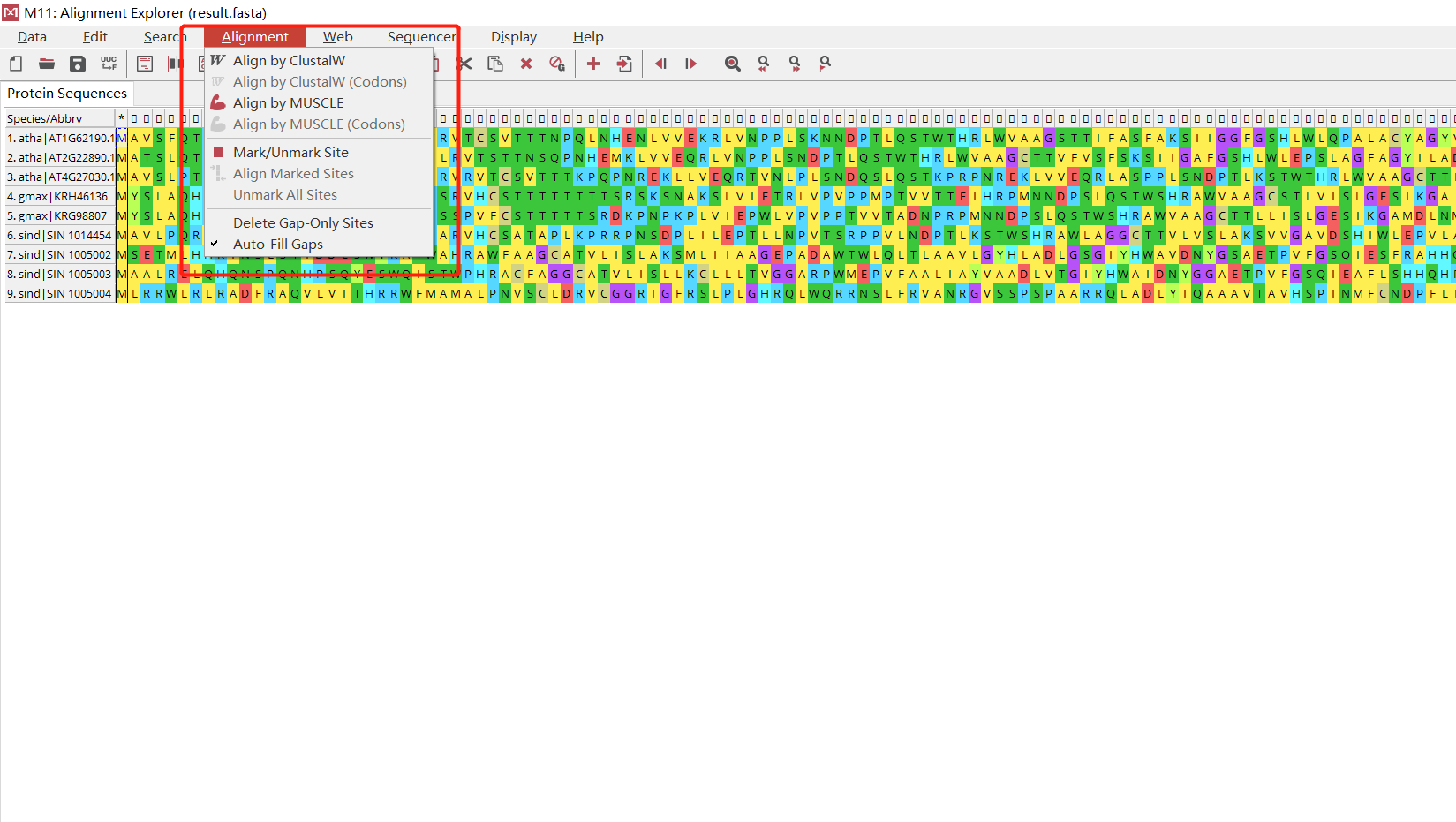
选择clust 或者 muscle
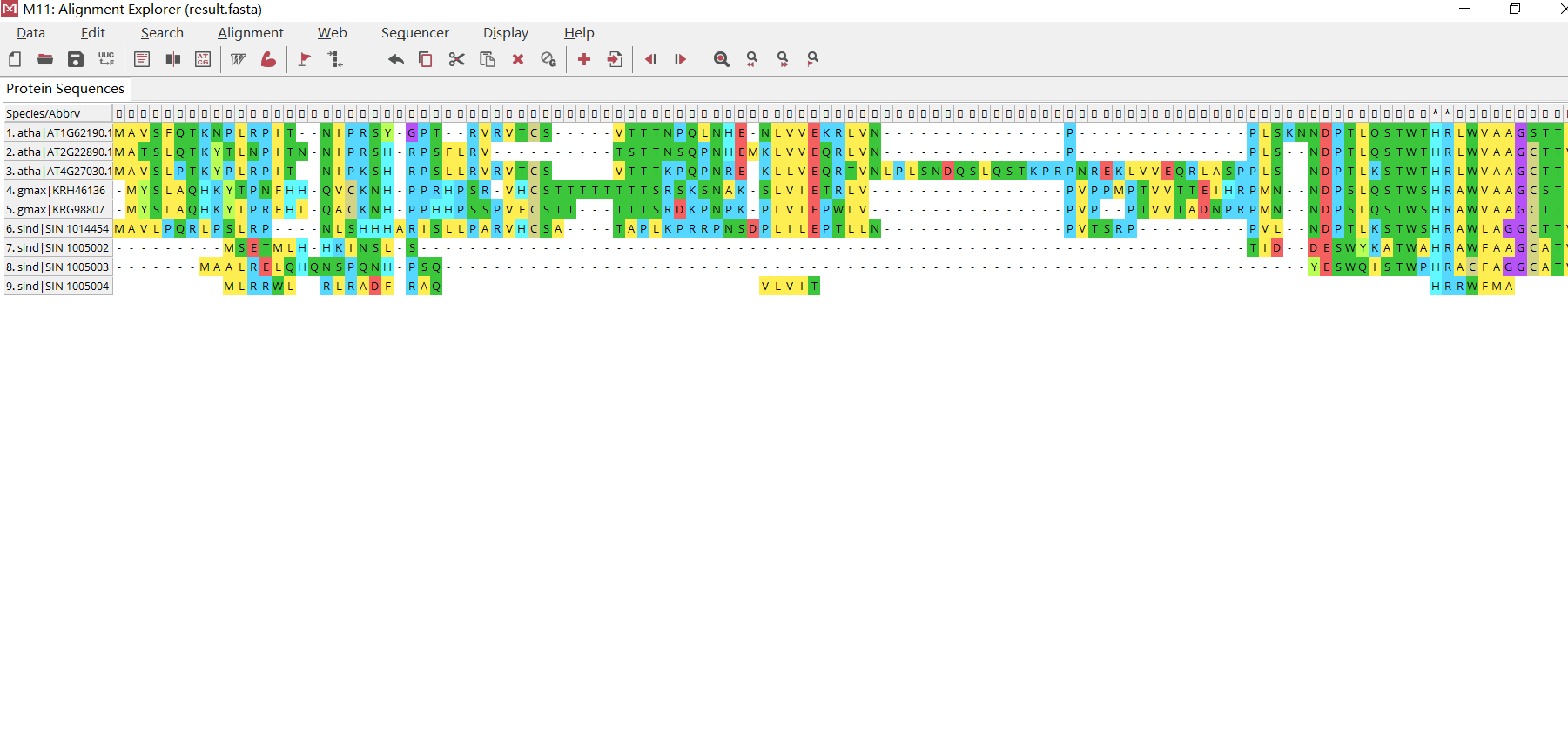
然后做进化树分析
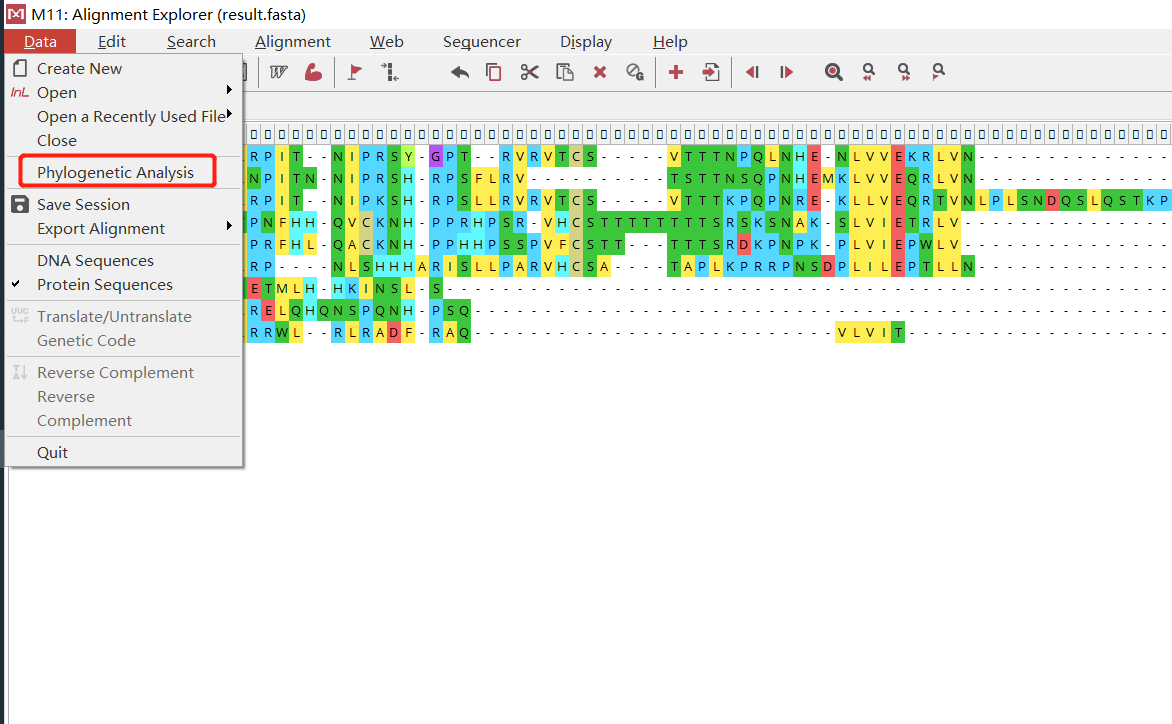
这里使用最大似然分析或者NJ法
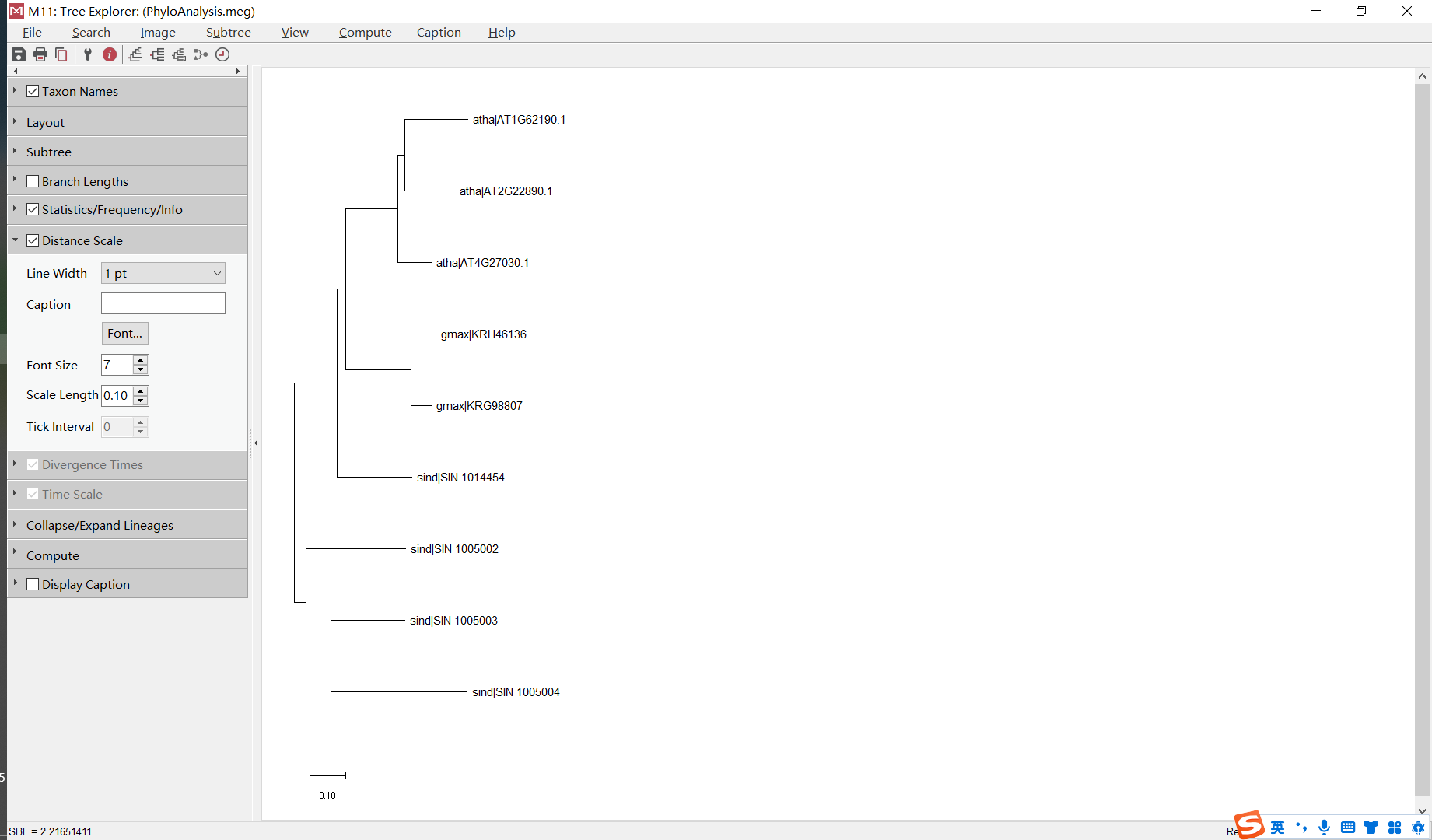
修改拓扑结构
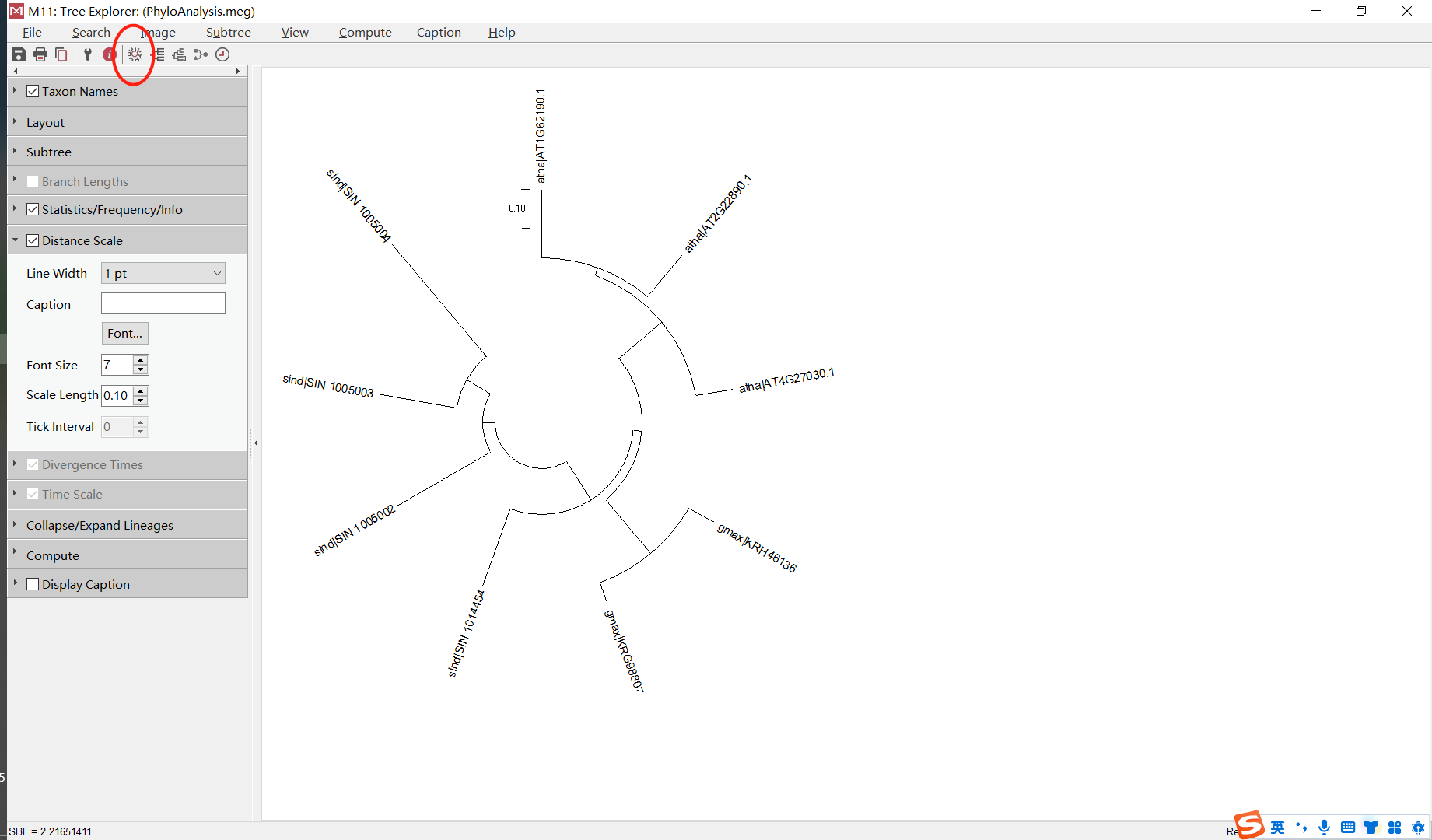
还可以调整颜色
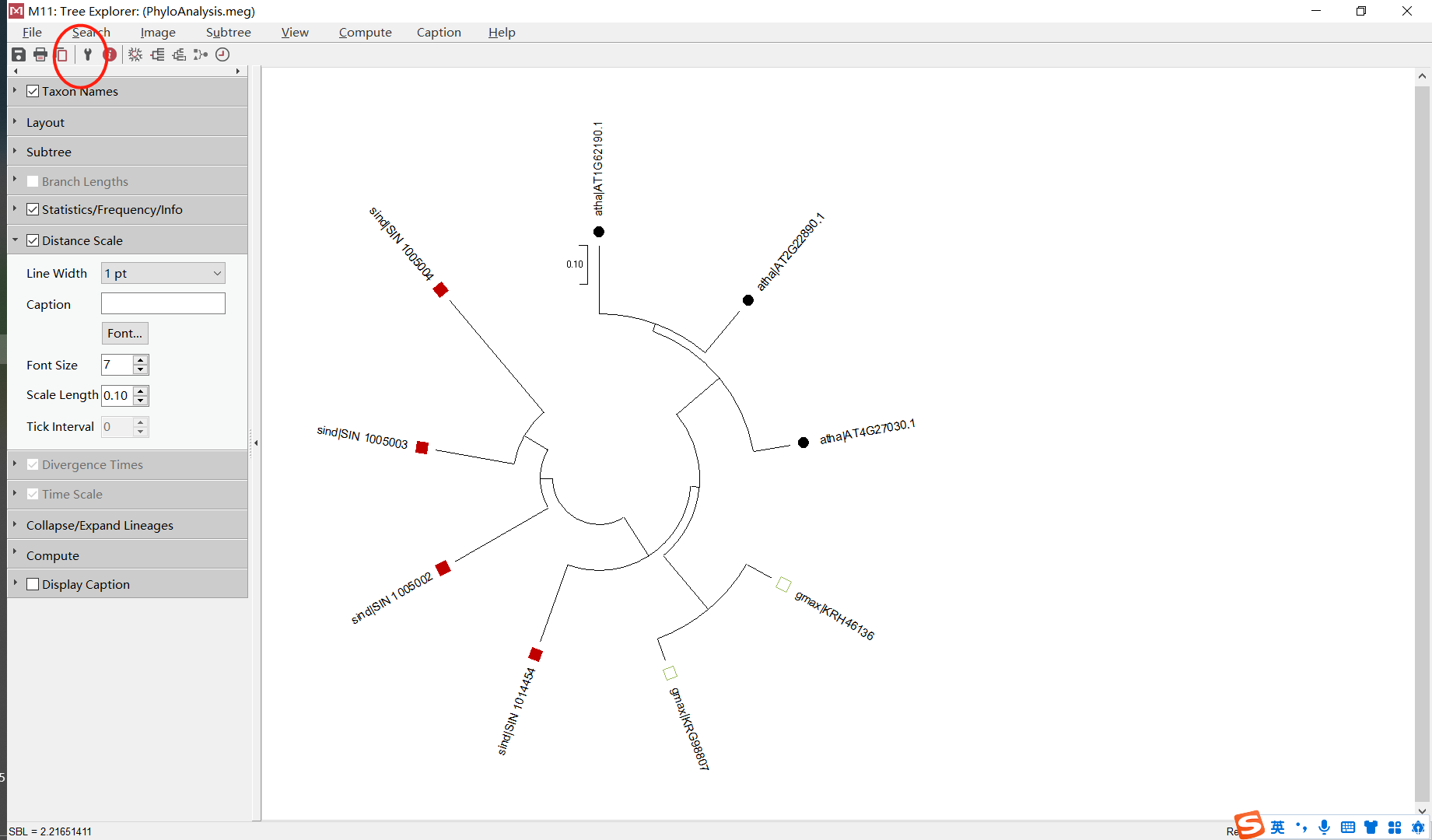
03 结果
进化树显示芝麻FAD4-like基因有4个拷贝。拷贝数是否与油脂含量高相关,需进一步验证。













